檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
核基因組突變是多種疾病的根源,而線粒體作為細胞内具有半自主功能的細胞器,擁有獨立的基因組,其基因組突變同樣與多種遺傳疾病密切相關。線粒體疾病通常累及多種組織器官,其中最為人熟知的包括Leigh綜合征和LHON(Leber遺傳性視神經病變)。Leigh綜合征的症狀包括發育遲緩、肌張力減退、運動和呼吸障礙等,而LHON則表現為視力喪失、中央暗點和視神經萎縮等問題。根據MITOMAP的統計,目前已驗證的線粒體緻病性突變有97個,其中點突變占比高達95%。然而,由于缺乏有效的點突變相關線粒體疾病小鼠模型,線粒體疾病的研究與治療開發受到了嚴重制約。
早期的小鼠模型主要通過化學誘導或遺傳工程構建[1],但這些方法操作複雜、成本高昂且對突變的精準控制較差,僅成功建立了極少數模型。近年來,研究人員成功開發了線粒體堿基編輯工具,可以對線粒體DNA實現C到T和A到G的編輯,例如DdCBEs和TALEDs。這些工具基于雙鍊DNA脫氨酶DddA蛋白[2, 3]。雖然已有研究者嘗試将這些工具應用于小鼠模型的構建,但其編輯效率尚不足以模拟人類線粒體疾病中高突變負荷的特征[4, 5]。此外,研究表明DdCBEs可能引發大量核基因組脫靶效應,這種非TALE依賴性的脫靶主要源于DddA蛋白的自組裝以及其與CTCF的相互作用[6]。因此,基于DddA的線粒體堿基編輯工具在應用中面臨核基因組脫靶的風險,難以直接建立線粒體突變與疾病表型之間的因果聯系。
針對這一挑戰,北京大學魏文勝課題組之前開發了mitoBEs,這是一種結合切口酶與單鍊DNA脫氨酶的新型線粒體堿基編輯工具,能夠實現線粒體DNA的C到T和A到G編輯。與DdCBEs和TALEDs相比,mitoBEs展現出卓越的鍊特異性和顯著降低的脫靶效應。得益于其雙向堿基編輯能力,mitoBEs能夠對大約87%的緻病線粒體突變進行精确建模[7]。
2025年1月22日,北京大學及昌平實驗室魏文勝課題組在Nature雜志在線發表了題為“Precise modelling of mitochondrial diseases using optimized mitoBEs”的研究論文。該研究報道了通過優化後的mitoBEs實現高效且精準地構建線粒體疾病小鼠模型的成果。利用優化版mitoBEs,研究團隊成功建立了具有高突變頻率的小鼠模型,這些模型表現出了與疾病相關的典型表型。此外,通過雜交實驗,還獲得了突變負荷達到100%以及僅含單堿基突變的精确小鼠模型。
為準确建立突變與疾病表型之間的直接聯系,消除堿基編輯工具的脫靶效應尤為重要。在利用mitoBEs進行建模時,需要将RNA編碼的mitoBEs注射到小鼠受精卵中。因此,該研究首先對RNA編碼的mitoBEs系統的脫靶效應進行了全面評估。結果表明,RNA編碼的mitoABE存在廣泛的轉錄組脫靶效應,而mitoCBE則表現出一定程度的依賴于APOBEC1蛋白的線粒體基因組脫靶效應。為了提高mitoBEs的精準性,該研究重點優化了脫氨酶。針對mitoABE,通過突變篩選發現,TadA8e-V106W-V28F能夠顯著降低轉錄組脫靶至背景水平(圖1)。針對mitoCBE,篩選了多種現有的胞嘧啶脫氨酶,并發現TadA衍生的胞嘧啶脫氨酶CBE6d在線粒體基因組上表現出的脫靶效應接近背景水平。基于這些優化成果,研究團隊将改進後的mitoBEs命名為mitoBEs v2,包括mitoABE v2和mitoCBE v2(圖1)。此外,該研究還系統性地評估了優化前後mitoBEs在核基因組上的脫靶效應,結果顯示,無論是優化前還是優化後的mitoBEs,均未在核基因組上引發明顯的脫靶效應,從而驗證了其在基因編輯中的安全性和可靠性。
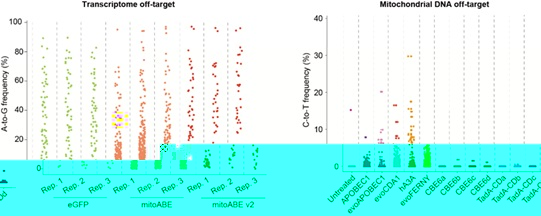
圖1 優化mitoBEs的編輯精準性
通過将85個人類緻病性線粒體DNA點突變與小鼠線粒體基因組進行同源性比對,研究确定了70個可編輯位點。進一步的細胞水平初步篩選成功實現了其中68個位點的編輯。比較發現,由環狀RNA(circRNA)編碼的mitoBEs v2相比于mRNA編碼的工具,具有更高的編輯效率。因此,研究團隊将circRNA編碼的mitoBEs v2注射至小鼠胚胎并進行移植,結果顯示mitoBEs v2在多種F0代小鼠模型中均實現了較高的編輯效率,其中mt-Nd5 A12784G F0小鼠模型的突變頻率高達82%(圖2)。此外,該研究系統性評估了F0代小鼠在線粒體基因組和核基因組中的脫靶效應,結果表明,在整個基因組範圍内未檢測到脫靶效應。這一發現表明,mitoBEs v2能夠構建遺傳背景幹淨的線粒體疾病小鼠模型。更重要的是,線粒體基因組的編輯結果在小鼠不同組織中表現出廣泛且持久的存在(圖2),并且能夠通過母系遺傳穩定傳遞。通過進一步雜交實驗,研究成功獲得了目标位點編輯效率達到100%以及僅含目标位點突變的mt-Nd5 A12784G小鼠模型。
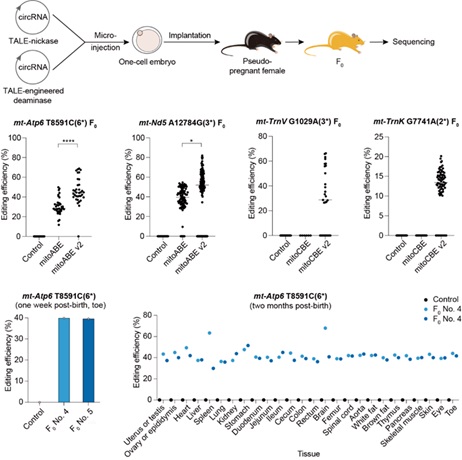
圖2 mitoBEs v2高效構建線粒體疾病小鼠模型,編輯結果廣泛且持久穩定于多組織
mt-Atp6 T8591C和mt-Nd5 A12784G分别對應人類線粒體緻病突變m.T9191C和m.A13379G,并分别導緻Leigh綜合征和LHON。研究團隊對突變率較高的F0代小鼠進行了疾病表型評估,結果顯示,mt-Atp6 T8591C小鼠表現出顯著的心髒功能障礙,與Leigh綜合征的臨床特征相符;mt-Nd5 A12784G小鼠則表現出類似LHON的視力障礙(圖3)。此外,研究還通過調整TALE結合位點,成功構建了僅含目标位點編輯的單堿基突變mt-Nd5 A12784G小鼠模型。這些研究結果充分證明了mitoBE v2在創建線粒體疾病小鼠模型方面的高效性和精準性,為深入探索線粒體疾病的緻病機制及開發新型治療策略提供了重要工具。
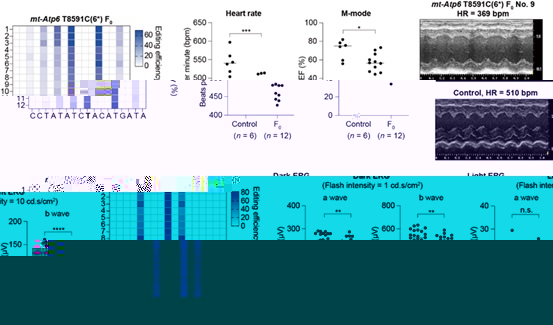
圖3 小鼠模型呈現出相應的線粒體疾病表型
北京大學博士後伊宗裔為該論文的共同通訊作者,昌平實驗室博士後張小雪為論文的第一作者,張雪、任紀武、李佳怡、魏曉旭和于瑩博士也為該研究做出了重要貢獻。本研究得到了昌平實驗室、國家自然科學基金、北大-清華生命科學聯合中心及中國博士後科學基金的資助。
文章鍊接: https://www.nature.com/articles/s41586-024-08469-8
參考文獻:
1. Stewart, J.B., Current progress with mammalian models of mitochondrial DNA disease. Journal of Inherited Metabolic Disease, 2021. 44(2).
2. Mok, B.Y., et al., A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature, 2020. 583(7817).
3. Cho, S.I., et al., Targeted A-to-G base editing in human mitochondrial DNA with programmable deaminases. Cell, 2022. 185(10).
4. Silva-Pinheiro, P., et al., A library of base editors for the precise ablation of all protein-coding genes in the mouse mitochondrial genome. Nature Biomedical Engineering, 2023. 7(5).
5. Cho, S.I., et al., Engineering TALE-linked deaminases to facilitate precision adenine base editing in mitochondrial DNA. Cell, 2024. 187(1).
6. Lei, Z.X., et al., Mitochondrial base editor induces substantial nuclear off-target mutations. Nature, 2022. 606(7915).
7. Yi, Z.Y., et al., Strand-selective base editing of human mitochondrial DNA using mitoBEs. Nature Biotechnology, 2024. 42(3).