檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
哺乳動物細胞核中的基因轉錄過程由三種RNA聚合酶複合物(Pol I、II、III)相互協同完成。其中,RNA聚合酶III(Pol III)不僅是細胞核内轉錄tRNA、5S rRNA、SINEs等短基因的核心機器[1],還參與DNA雙鍊斷裂的同源重組修複[2],同時在細胞質中通過轉錄富含A-T的DNA病毒模闆來觸發天然免疫[3, 4]。與其他兩種RNA聚合酶主要定位于細胞核内發揮功能不同,Pol III在細胞質中作為病毒DNA的感知器同樣具有轉錄活性,但細胞質和細胞核内Pol III的組裝和分布調控機制尚不清楚。
近日,beat365官方网站、北大-清華生命科學聯合中心季雄課題組在Cell Reports雜志上在線發表了題為“Subcellular localization shapes the fate of RNA polymerase III”的研究論文。研究中對Pol III的組裝和降解進行了分析,利用蛋白快速降解系統、活細胞鄰近标記的蛋白質組和活細胞熒光次序标記的實時成像系統,發現Pol III核心催化亞基RPC1的降解會導緻細胞核内Pol III複合物的快速降解,而在細胞質中則會形成去組裝的Pol III中間亞複合體,這些中間體不會被降解,并且在RPC1蛋白水平恢複時重新組裝成Pol III複合物并進入細胞核。研究還發現,蛋白酶體和分子伴侶HSP90在細胞核中對Pol III的穩态調控起着關鍵作用,并推測Pol III由細胞質進入細胞核是一個單向過程,即Pol III首先在細胞質中組裝為核心複合物,然後在細胞核中成熟為完整的全酶從而執行轉錄功能。

為了研究Pol III的生命周期 (life cycle),研究者在小鼠胚胎幹細胞(mESC)中對内源亞基進行基因編輯。其中,一個亞基采用蛋白瞬時降解标簽(Degron),另一個不同亞基采用HaloTag标簽或TurboID标簽進行活細胞标記。當誘導Degron标記的亞基發生降解時,通過實時熒光标記成像檢測發現Pol III的其他亞基在細胞核内逐漸減少,而在細胞質中除了被誘導降解的亞基外,其他亞基的蛋白水平變化不大。有趣的是,發現亞基RPC3主要定位在細胞核,即使細胞核内的Pol III全酶降解後,RPC3仍然滞留在核内,并與RPC6形成穩定的亞複合物。
為了确定細胞核内亞基減少是否是由于Pol III去組裝後被運出細胞核,研究者對細胞進行了出核抑制劑處理,并發現這并未影響Pol III在細胞核内的亞基減少。然而,當采用蛋白酶體抑制劑處理細胞時,細胞核内出現了Pol III亞基的積累現象。同時,研究者使用鄰近标記(RPC2-TurboID)的方法嘗試捕獲參與Pol III命運決定的蛋白質,當RPC1被誘導降解時,通過比較降解前後的蛋白質組差異,發現輔助蛋白質去折疊的分子伴侶和參與蛋白質降解的組分在細胞核内富集。這些結果表明,細胞核内的Pol III最終通過細胞核内的蛋白酶體降解而不是被運送到細胞質中進行處理。
随後,研究者利用HaloTag标記RPC2的不同顔色形成時序标記,以區分新舊蛋白質,并根據标記的時間順序區分它們。通過人為控制RPC1的降解和蛋白表達的恢複,研究者發現,當RPC1表達恢複後,細胞質中的新舊RPC2仍然可以重新組裝成功能完整的Pol III複合物,并被運送到細胞核内發揮功能。
綜上所述,該研究利用蛋白快速降解和活細胞實時标記系統發現,RNA聚合酶III在細胞質中通過分子伴侶蛋白的協助進行組裝,部分留在細胞質中發揮與天然免疫相關的功能,其餘部分進入細胞核内發揮轉錄功能,并最終在細胞核内經過蛋白酶體系統降解。
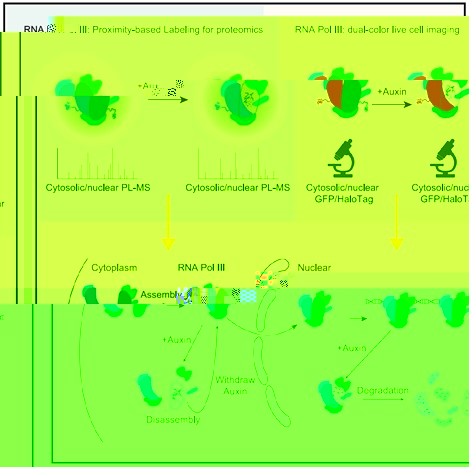
圖文摘要. 通過瞬時降解聯合活細胞标記的方法,揭示了亞細胞定位在決定RNA聚合酶III命運中的作用。
beat365官方网站、北大-清華生命科學聯合中心季雄研究員為該論文的通訊作者。beat365博士後田凱(已出站)、博士後王蕊和黃捷為該論文的共同第一作者。beat365官方网站王輝博士(已出站,現為四川農大副教授)為該工作提供了重要幫助。該工作得到北大-清華生命科學聯合中心、啟東創新基金、細胞增殖與分化教育部重點實驗室、科技部國家重點研發計劃、國家自然科基金和中國博士後科學基金的資助。感謝北京大學鳳凰工程多個儀器平台對本項目的大力支持。
季雄課題組長期從事RNA聚合酶非經典功能調控研究。主要集中在RNA聚合酶亞基未知功能調控、分子探針和非模式生物等方向,近5年成果發表在Cell(2023)、Molecular Cell(2022,2023)、Genome Biology(2020,2022)、Nature Communications (2022)、Nucleic Acids Research (2023)、Cell Reports (2023)、Cell Discovery (2020)、CMLS(2022)、iScience(2022)、Transcription (2023)、STAR Protocol(2023a, 2023b)等雜志上,為選擇性基因表達調控提供新的假說。現因發展需要,招聘博士後1-2名。
原文鍊接: https://doi.org/10.1016/j.celrep.2023.112941
參考文獻
1. White, R. J. (2011). Transcription by RNA polymerase III: more complex than we thought. Nature Reviews Genetics, 12(7), 459-463.
2. Liu, S., Hua, Y., Wang, J., Li, L., Yuan, J., Zhang, B., ... & Kong, D. (2021). RNA polymerase III is required for the repair of DNA double-strand breaks by homologous recombination. Cell, 184(5), 1314-1329.
3. Chiu, Y. H., MacMillan, J. B., & Chen, Z. J. (2009). RNA polymerase III detects cytosolic DNA and induces type I interferons through the RIG-I pathway. Cell, 138(3), 576-591.
4. Ablasser, A., Bauernfeind, F., Hartmann, G., Latz, E., Fitzgerald, K. A., & Hornung, V. (2009). RIG-I-dependent sensing of poly (dA: dT) through the induction of an RNA polymerase III–transcribed RNA intermediate. Nature immunology, 10(10), 1065-1072.