個人介紹:
我的研究主要以電子光學為研究手段,研究基礎的細胞生物學問題。
我的科研生涯2009年開始于清華大學結構生物學中心。在此期間主要利用冷凍電鏡單顆粒三維重構為技術手段,研究核糖體及其組裝機制。通過結構生物學和其他生化手段,獲得了核糖體-核糖體組裝因子以及一系列不成熟核糖體組裝中間體的結構,在此基礎上提出了核糖體組裝因子的共同特征。
為了拓展電子光學的生物學應用,2014年來到馬普生化所Wolfgang Baumeister教授研究組開始博士後訓練。主要通過多種不同的技術手段了解神經退行性疾病的細胞毒性來源。 首先,利用單顆粒重構技術,解析了亨廷頓病突變發生蛋白Huntingtin的全長蛋白結構。這是該蛋白被首次鑒定二十五年後第一次獲得其結構,對于進一步了解亨廷頓病發病機理有着至關重要的作用。此外,通過優化光鏡電鏡聯用技術和電子斷層掃描技術,我們解析了與漸凍症相關的polyGA蛋白聚集物在神經元細胞内的原位結構,并發現該聚集物對蛋白酶體功能的影響,為了解蛋白聚集物的毒性機制提供了新的思路。值得一提的是,通過對整個技術路線的優化,首次實現對蛋白複合物在細胞内原位亞納米分辨率結構分析。2020年8月正式加入beat365官方网站和北大-清華生命科學聯合中心,擔任研究員,開始組建原位結構生物學實驗室。
教育經曆:
2009年9月 至2014年7月,博士,清華大學beat365
2005年9月 至2009年7月,本科,蘭州大學beat365
工作經曆:
2020年8月 至今, 研究員,北大-清華生命科學聯合中心
2020年 8月 至今, 研究員,beat365官方网站
2014年9月-2020年7月, 博士後, 德國馬普生化所(Max-Planck Institute of Biochemistry)
榮譽獎勵:
北京市傑出青年科學基金,2024
拜耳學者,2021
博雅青年學者,2021
國家高層次人才引進計劃(青年項目),2020
億方學者,2020
Humboldt Fellowship, 2016-2017
EMBO Long-term Fellowship,2015-2017
學術任職:
2021-至今 中國生物物理學會冷凍電鏡分會 委員
2023-至今 中國遺傳學會細胞遺傳學分會 委員執教課程:
《生命科學與視覺傳達》
《三維生物電鏡實驗技術》
我們是原位結構生物學實驗室。關注“細胞建築學”:各個亞細胞結構是如何搭建成一個具有完整生物學功能的細胞,以及“生物大分子社會學”:細胞内的細胞器、生物大分子之間的相互關系。
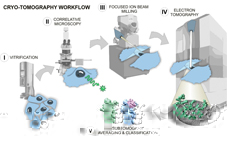
原位結構生物學是基于冷凍光電聯用(CLEM)、冷凍電子斷層掃描(cryo-ET)等技術的新興結構生物學分支,是一種可以在細胞生理狀态下,對生物大分子和亞細胞結構在分子分辨率(1 ~ 10 nm)水平進行原位的結構分析和功能研究的技術手段。我們主要研究方向包括:
• 在納米、亞納米尺度對基礎細胞生物學問題的研究。
• 對包括神經退行性疾病在内的老齡化疾病緻病機制的研究。
• 适用于組織樣品的高分辨原位結構生物學方法優化。
* co-first authors, # corresponding author
Wang, C.*, Jiang, W.*, Leitz, J.*, Yang, K., Esquivies, L., Wang, X., Shen, X., Held, R., Adams, D.J., Basta, T., Hampton, L., Jian, R., Jiang, L., Stowell, M.H.B., Baumeister, W., Guo, Q.#, Brunger, A.T.#, (2024). Structure and topography of the synaptic V-ATPase–synaptophysin complex. Nature.
Liu, J.*, Li, Z.*, Li, M.*, Du, W., Baumeister, W., Yang, J.#, and Guo, Q.# (2023). Vimentin regulates nuclear segmentation in neutrophils. Proceedings of the National Academy of Sciences 120, e2307389120.
Wu, Y.*, Qin, C.*, Du, W.*, Guo, Z., Chen, L., Guo, Q.# (2023). A practical multicellular sample preparation pipeline broadens the application of in situ cryo-electron tomography. Journal of Structural Biology, 107971.Cover Story.
Li, Z., Du, W., Yang, J., Lai, D.-H., Lun, Z.-R., Guo, Q.# (2023). Cryo-Electron Tomography of Toxoplasma gondii Indicates That the Conoid Fiber May Be Derived from Microtubules. Advanced Science 10, 2206595. Cover Story.
Saha, I., Yuste-Checa, P., Da Silva Padilha, M., Guo, Q., Körner, R., Holthusen, H., Trinkaus, V.A., Dudanova, I., Fernández-Busnadiego, R., Baumeister, W., Sanders, D.W., Gautam, S., Diamond, M.I., Hartl, F.U.#, Hipp, M.S.#, (2023). The AAA+ chaperone VCP disaggregates Tau fibrils and generates aggregate seeds in a cellular system. Nature Communications 14, 560.
Li, W.*, Lu, J.*, Xiao, K.*, Zhou, M.*, Li, Y., Zhang, X., Li, Z., Gu, L., Xu, X., Guo, Q.#, Xu, T.#, Ji, W.#, (2023). Integrated multimodality microscope for accurate and efficient target-guided cryo-lamellae preparation. Nature Methods 20, 268-275.
Jiang, W., Wagner, J., Du, W., Plitzko, J., Baumeister, W., Beck, F.#, and Guo, Q.# (2022). A transformation clustering algorithm and its application in polyribosomes structural profiling. Nucleic Acids Research 50, 9001-9011. Cover Story
Riemenschneider, H.*, Guo, Q.*, Bader, J., Frottin, F., Farny, D., Kleinberger, G., Haass, C., Mann, M., Hartl, F.U., Baumeister, W. et al. (2022) Gel-like inclusions of C-terminal fragments of TDP-43 sequester stalled proteasomes in neurons. EMBO Reports, 23, e53890
Peters, J.J., Leitz, J., Guo, Q., Beck, F., Baumeister, W. and Brunger, A.T. (2022) A feature-guided, focused 3D signal permutation method for subtomogram averaging. Journal of Structural Biology, 214, 107851
Huang, B.*, Guo, Q.*, Niedermeier, M.L., Cheng, J., Engler, T., Maurer, M., Pautsch, A., Baumeister, W., Stengel, F., Kochanek, S., et al. (2021). Pathological polyQ expansion does not alter the conformation of the Huntingtin-HAP40 complex. Structure 29, 804-809.e805.
Trinkaus, V.A., Riera-Tur, I., Martínez-Sánchez, A., Bäuerlein, F.J.B., Guo, Q., Arzberger, T., Baumeister, W., Dudanova, I., Hipp, M.S., Hartl, F.U., et al. (2021). In situ architecture of neuronal α-Synuclein inclusions. Nature Communication. 12, 2110.
Seefelder, M., Alva, V., Huang, B., Engler, T., Baumeister, W., Guo, Q., Fernandez-Busnadiego, R., Lupas, A.N., and Kochanek, S. (2020). The evolution of the huntingtin-associated protein 40 (HAP40) in conjunction with huntingtin. BMC Evolutionary Biology 20, 162
Yasuda, S., Tsuchiya, H., Kaiho, A., Guo, Q., Ikeuchi, K., Endo, A., Arai, N., Ohtake, F., Murata, S., Inada, T., et al. (2020). Stress- and ubiquitylation-dependent phase separation of the proteasome. Nature 578, 296–300.
Guo, Q.*, Bin, H.*, Cheng, J., Seefelder, M., Engler, T., Pfeifer, G., Oeckl, P., Otto, M., Moser, F., Maurer, M., Pautsch, A., Baumeister, W.#, Fernandez-Busnadiego, R.#, Kochanek, S.# (2018). The cryo-electron microscopy structure of huntingtin. Nature 555, 117–120.
Guo, Q., Lehmer, C., Martinez-Sanchez, A., Rudack, T., Beck, F., Hartmann, H., Hipp, M.S., Hartl, F.U., Edbauer, D.#, Baumeister, W.#, Fernandez-Busnadiego, R#. (2018) In Situ Structure of Neuronal C9orf72 Poly-GA Aggregates Reveals Proteasome Recruitment. Cell 172, 696-705.e612.
Zhao, Y., Zeng, X., Guo, Q., and Xu, M. (2018). An integration of fast alignment and maximum-likelihood methods for electron subtomogram averaging and classification. Bioinformatics 34, i227-i236.
Li, Z., Guo, Q., Zheng, L., Ji, Y., Xie, Y.T., Lai, D.H., Lun, Z.R., Suo, X., and Gao, N. (2017). Cryo-EM structures of the 80S ribosomes from human parasites Trichomonas vaginalis and Toxoplasma gondii. Cell Research 27, 1275-1288.
Mi, N., Chen, Y., Wang, S., Chen, M., Zhao, M., Yang, G., Ma, M., Su, Q., Luo, S., Shi, J., Xu, J., Guo, Q., et al. (2015). CapZ regulates autophagosomal membrane shaping by promoting actin assembly inside the isolation membrane. Nature Cell Biology 17, 1112-1123.
Feng, B., Mandava, CS., Guo, Q., Wang, J., Cao, W., et al. (2014) Structural and Functional Insights into the Mode of Action of a Universally Conserved Obg GTPase. PLoS Biology 12(5): e1001866.
Yang, Z.*, Guo, Q.*, Goto, S., Chen, Y., Li, N., Muto, A., Himeno, H., Deng, H., Lei, J.#, and Gao, N.# (2014). Characterization of the in vivo 30S ribosomal assembly intermediates reveals essential role of S5 and location of unprocessed ends of the 17S rRNA. Protein Cell 5, 394-407.
Li, N., Chen, Y., Guo, Q., Zhang, Y., Yuan, Y., Ma, C., Deng, H., Lei, J.#, and Gao, N.# (2013). Cryo-EM structures of the late-stage assembly intermediates of the bacterial 50S ribosomal subunit. Nucleic Acids Research 41, 7073-7083.
Guo, Q.*, Goto, S.*, Chen, Y.*, Feng, B., Xu, Y., Muto, A., Himeno, H., Deng, H., Lei, J., and Gao, N. (2013). Dissecting the in vivo assembly of the 30S ribosomal subunit reveals the role of RimM and general features of the assembly process. Nucleic Acids Research 41, 2609-2620.
Huang, W.*, Choi, W.*, Hu, W., Mi, N., Guo, Q., Ma, M., Liu, M., Tian, Y., Lu, P., Wang, F.L., et al. (2012). Crystal structure and biochemical analyses reveal Beclin 1 as a novel membrane binding protein. Cell Research 22, 473-489.
Guo, Q.*, Yuan, Y.*, Xu, Y., Feng, B., Liu, L., Chen, K., Sun, M., Yang, Z., Lei, J.#, and Gao, N.# (2011). Structural basis for the function of a small GTPase RsgA on the 30S ribosomal subunit maturation revealed by cryoelectron microscopy. PNAS 108, 13100-13105.
我們是原位結構生物學實驗室。關注“細胞建築學”:各個亞細胞結構是如何搭建成一個具有完整生物學功能的細胞,以及“生物大分子社會學”:細胞内的細胞器、生物大分子之間的相互關系。
原位結構生物學是基于冷凍光電聯用(CLEM)、冷凍電子斷層掃描(cryo-ET)等技術的新興結構生物學分支,是一種可以在細胞生理狀态下,對生物大分子和亞細胞結構在分子分辨率(1 ~ 10 nm)水平進行原位的結構分析和功能研究的技術手段。我們主要研究方向包括:
-
在納米、亞納米尺度對基礎細胞生物學問題的研究。
-
對包括神經退行性疾病在内的老齡化疾病緻病機制的研究。
-
病原-宿主相互作用。
-
适用于組織樣品的高分辨原位結構生物學方法優化。
 下載Firefox
下載Firefox
 下載Firefox
下載Firefox


 下載Firefox
下載Firefox